-
한국화학연구원 코로나19 치료용 항체.진단 관련 연구결과 발표

너무나 기다린 소식입니다.
코로나는 코리아를 이길수없습니다.
보도자료
배포일 2020년 3월 4일(수)
https://www.krict.re.kr/news/bbs/view/tableid/news/id/92318
보도자료 | 한국화학연구원
□ 전 세계 연구진이 코로나19에 대항할 수 있는 연구를 진행하고 있는 가운데, 국내 연구진이 코로나19 치료용 항체 및 진단 개발에 활용할 수 있는 연구 결과를 발표했다. ◯ 한국화학연구원은 CEVI(신종 바이러스) 융합연구단은 기존에 알려진 사스 중화항체 2개와 메르스 중화항체 1개가 코로나19 스파이크 단백질에 결합할 수 있다는 결과를 예측했다. ◯ 스파이크 단백질은 코로나 바이러스가 세포 내로 침입할 때 활용되는 단백질이다. 연구진은 이 스파이크
www.krict.re.kr

□ 전 세계 연구진이 코로나19에 대항할 수 있는 연구를 진행하고 있는 가운데,
국내 연구진이 코로나19 치료용 항체 및 진단 개발에 활용할 수 있는 연구 결과를 발표했다.
◯ 한국화학연구원은 CEVI(신종 바이러스) 융합연구단은 기존에 알려진
사스 중화항체 2개와 메르스 중화항체 1개가 코로나19
스파이크 단백질에 결합할 수 있다는 결과를 예측했다.
◯ 스파이크 단백질은 코로나 바이러스가 세포 내로 침입할 때 활용되는 단백질이다.
연구진은 이 스파이크 단백질에 결합할 수 있는 항체를 예측한 것이다.
항체는 인체에 침입하는 바이러스를 무력화하기 위해 우리 몸의 면역반응이 만든 일종의 무기다.
◯ CEVI 융합연구단은 코로나19의 유전체 분석을 통해 사스 바이러스와의 유사성을 확인했고,
기존의 사스와 메르스 중화항체가 코로나19에 결합할 수 있는지 생물정보학 분석을 통해 예측했다.
◯ 연구진은 긴급히 연구를 수행하기 위해 이미 ‘bioRxiv’에 공개된 코로나19 스파이크 단백질의
구조 정보 파일을 저자로부터 전달받아 예측 연구를 수행했다.
◯ 그 결과, 기존의 사스 중화항체 2개, 메르스 항체 1개가
코로나19 스파이크 단백질에 결합할 수 있다는 결과를 예측했다.
이 같은 연구결과는 코로나19 치료용 항체 및 백신 개발을 앞당기는 데 기여할 것으로 기대된다.
◯ 연구진은 이를 생물학 분야 아카이브인 ‘bioRxiv’에 2월 23일 투고했고,
‘bioRxiv’는 2월 27일 이를 공개했다. 현재 코로나19 관련 주요
연구결과는 ‘bioRxiv’에 빠르게 먼저 공개된 후 과학저널에 게재되고 있다.
□ 또한 CEVI 융합연구단은 2월 17일 질병관리본부로부터
코로나19 바이러스 분리주를 분양받아 한국화학연구원 생물안전 3등급
시설에서 신속한 배양을 통해 코로나19 바이러스 RNA를 확보했다.
◯ 연구진은 이를 이용해 해외에서 공개된 코로나19 바이러스 검출용 프라이머·프로브 세트*의 민감도를 비교했다.
프라이머는 특정 유전자 합성의 시작점이 되는 짧은 유전자 서열이고,
프로브는 특정 유전자의 증폭을 실시간으로 판독할 수 있는 형광이 표지된 짧은 유전자 서열이다.
*프라이머프로브 세트 민감도 비교 분석표
(출처: doi: https://doi.org/10.1101/2020.02.25.964775)
※ 3 반복 실험 중 1회 양성 신호(Ct 값 : 43)
※ 민감도는 Ct 값이 상대적으로 낮을수록 우수
◯ 그 결과, 동일 조건*에서 코로나19 바이러스 N 유전자 검출용은
미국 질병통제센터의 2019-nCoV_N2, N3, 일본 국립감염병연구소의
NIID_2019-nCOV_N 프라이머·프로브 세트가 민감한 것으로 확인했다.
*동일조건에서 프라이머·프로브 염기서열만을 비교한 것으로 실제 사용 키트의 성능비교는 아니며,
각 키트의 조성 및 시약에 따라 차이가 발생할 수 있음
◯ 또한 RdRp/Orf1 유전자 검출용은 중국 질병예방통제센터의
ORF1ab 프라이머프로브 세트가 민감한 것으로 확인했다.
◯ 전 세계 코로나19 검출용 주요 프라이머·프로브 세트의 민감도를 비교한 건 이번이 처음으로,
보다 민감한 실시간 유전자 증폭 기반의 분자진단키트를 개발하는 데 활용될 것으로 기대된다.
◯ 이번 연구결과는 2월 25일 바이오아카이브 ‘bioRxiv’에 투고됐으며, 2월 27일 공개됐다.
◯ 한국화학연구원 이미혜 원장은 “코로나19 바이러스 진단기술,
백신, 치료제 개발을 위해 적극적인 지원을 아끼지 않겠다”면서
“앞으로도 국민 건강과 밀접한 감염병 해결을 위한 연구에 힘쓰겠다”고 말했다.
◯ 한국화학연구원 CEVI 융합연구단 김범태 단장은 “이번 코로나19 바이러스 대응을 위해
그동안 구축한 융합연구 역량을 총동원하겠다”고 밝혔다.
□ CEVI 융합연구단은 한국화학연구원을 중심으로 총 8개 정부출연연구기관*이
신종 바이러스 진단, 백신, 치료제 및 확산방지 기술 개발 연구를 수행하고 있다.
* 참여기관 : 한국건설연구원, 한국과학기술정보연구원, 한국기초과학지원연구원,
한국식품연구원, 한국표준과학연구원, 한국한의학연구원, 안전성평가연구소
◯ CEVI 융합연구단은 2월 17일 질병관리본부로부터 코로나19
바이러스 분리주를 분양받아 진단기술, 백신 및 치료제 개발에 활용하고 있다.
◯ CEVI 융합연구단 바이러스 진단팀은 코로나19 바이러스
항원 진단용 신속진단기술 개발 연구를 수행하고 있고, 바이러스 백신팀은 코로나19 바이러스
특성연구, 감염동물제작 및 백신연구를 진행하고 있다. 또 바이러스
치료제팀은 한국파스퇴르연구소와 함께 치료제 재창출 연구에 착수했다.
◯ 한편, 이번 연구는 과학기술정보통신부의 국가과학기술연구회
융합연구단사업과 한국연구재단의 국민생활안전 긴급대응연구사업의 지원으로 수행되고 있다.
연구 관련 이미지
1. 신종바이러스(CEVI) 융합연구단 조직도
2. 코로나19 바이러스 스파이크 단백질의 3차원 구조(A)와 사스 중화항체 (B) 및 메르스 중화항체(C) 결합 예측
출처: doi: https://doi.org/10.1101/2020.02.22.951178
Spike protein binding prediction with neutralizing antibodies of SARS-CoV-2
Coronavirus disease 2019 (COVID-19) is a new emerging human infectious disease caused by Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2, also previously known as 2019-nCoV), originated in Wuhan seafood and animal market, China. Since December
www.biorxiv.org

코로나19 바이러스 스파이크 단백질의 3차원 구조(A)와
사스 중화항체 (B) 및 메르스 중화항체(C) 결합 예측

출처: doi: https://doi.org/10.1101/2020.02.22.951178
Spike protein binding prediction with neutralizing antibodies of SARS-CoV-2
Coronavirus disease 2019 (COVID-19) is a new emerging human infectious disease caused by Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2, also previously known as 2019-nCoV), originated in Wuhan seafood and animal market, China. Since December
www.biorxiv.org
위 사이트를 한글로 번역하여 올려드립니다.
모든 자료 출처는 명시되어있는 주소에 나와 있는 자료를 올린겁니다.
https://papago.naver.net/website
네이버 파파고
번역을 부탁해 파파고
papago.naver.net
네이버파파고에서 위 사이트 주소를 입력하고 번역하여 포스팅하였습니다.
SA-CoV-2실험실 확인을 위한 프라이머-프로브 세트의 비교 분석
https://www.biorxiv.org/content/10.1101/2020.02.25.964775v1.full
추상적인
코로나 바이러스 질환은 2019년(COVID-19)은 심각한 급성 호흡기 증후군 코로나 바이러스 2(과거에는 2002-EC6으로도 알려져 있음)에 의해 발생한 새로운 인간 전염병이다. 발병 2개월 이내에 전 세계적으로 8만건 이상의 COVID-19사례가 확인되었다. 인간 대 인간 전염은 쉽게 일어나고 감염은 빠르게 증가하고 있기 때문에, 전 세계적인 발병을 막기 위해서는 민감하고 빠른 진단이 필수적이다. 최근, 세계 보건 기구는 이전에 중국, 독일, 홍콩, 일본, 태국, 미국에서 개발된 사스-CO-2에 대한 다양한 입문서와 조사 세트를 발표했다. 본 연구에서는 N유전자에 대한 7개의 프라이머-프로브 세트와 Orf1유전자에 대한 3개의 프라이머-프로브 세트 중에서 SA-CoV-2RNA를 검출하는 능력을 비교하였습니다. 비교 분석 결과에 따르면 미국의 '2014-CCEW_N2, N3'과 중국의 'OR17/G3'가 각각 N과 Orf1유전자에 대해 가장 민감한 문제 세트이다. 따라서 SARS-CoV-2의 민감하고 신뢰할 수 있는 실험실 확인을 위해 ORF10탭(중국), 2002-VGA_N(미국)및 NIID_2019-ECOVN(일본)세트의 적절한 조합을 선택해야 한다.
도입
2019년 12월 31일 세계 보건 기구(WHO)에 처음 통보된 현 코로나 바이러스 질병(COVID-19)의 발병은 2020년 2월 23일 현재 28개국에서 확인된 7만 811건을 포함한다[1]. 대다수의 COVID-19환자들은 폐렴을 앓았고 열과 기침을 포함한 증상을 보였다[2,3]. 원인이 되는 새로운 코로나 바이러스의 게놈 순서는 2020년 1월 12일부터 모든 GISAID(InfluenzaData)공유에 관한 글로벌 이니셔티브를 통해 공유되었다. 새로운 코로나 바이러스(CoV)의 시퀀스는 중증 급성 호흡기 증후군 관련 코로나 바이러스(SARSr-COV)와 유사하며 바이러스는 SAR2를 SARSCoV와 같은 입력 수용체로 사용한다[4-6]. 바이러스의 분류에 관한 국제 위원회의 코로나 바이러스 연구 그룹[7]은 바이러스를 사스-코발트-2로 지정했다.
COVID-19의 분자 진단은 중국, 독일, 홍콩, 일본, 태국, 미국의 기관들이 제안한 프라이머와 탐침이 WHO를 통해 게시된 1단계 정량적 RT-PCR(CMT-PCR)을 통해 수행되고 있습니다[8-10]. 2020년 2월 13일부터 중국 후베이성의 COVID-19사례를 식별하기 위해 CT스캔을 포함한 임상 진단 방법이 사용됩니다[11]. SRT-PCR분석은 SARS-CoV및 MERS-COV[12-15]와 같은 호흡기 감염 바이러스를 탐지하는 골드 표준 방법의 역할을 했지만, 현재의 TDT-PCR분석에서는 SARS-CoV-2의 대상을 측정하는 데 몇가지 주의 사항이 있습니다. 첫째, SA-CoV-2와 SA-CoV의 높은 유사성으로 인해 프라이머-프로브 세트가 교차할 수 있습니다. 둘째, 분석의 민감도는 입원 후 초기에 의심 환자를 확인하기에 충분하지 않을 수 있다. 실제로 최초 프레젠테이션에서 양성 CT스캔 결과와 음성 RT-PCR결과가 보고되었습니다[16]. 분자 진단의 성능은 프라이머, 탐침 및 시약에 따라 달라질 수 있습니다. SA-CoV-2의 분자 진단을 위한 현재의 PCR-PCR분석의 비교 결과는 없습니다.
본 연구에서는 이전에 보고된 SA-CoV-2의 경로/Orf1및 N지역을 대상으로 한 기본 프로브 세트를 사용하여 VTT-PCR분석을 수행했습니다. 이것은 SARS-CoV-2의 실험실 확인을 위한 다양한 프라이머-프로브 세트의 첫번째 비교 분석이다.
자재와 방법
ofCR의 프라이머 정보
SARS-COV-2의 실험실 확인 비교 분석을 위해, 질병 관리 예방 센터(CentersforDiseaseControlandPrevention, CDC)(미국), Charite–Virology베를린 연구소(Germany, Germany, Germany)의 6개 국가 기관의 염기 서열 정보를 바탕으로 10개의 프라이머-균군을 선정했다. 홍콩 대학교(홍콩), 국립 전염병 연구소, 바이러스학 III(일본), 중국 질병 통제 예방 센터(중국), 국립 보건 연구소(태국). 모든 DNA올리고 널 로베(대전)에서 합성되었다. 프라이머-primer-probe세트의 순서와 바이러스 RNA(GenBankMN908947.3)에서의 세트 위치는 그림 1과 표 1에 나열되어 있습니다. 10개 세트 중 7개는 N유전자에서 파생되었고, 나머지 3개 세트는 Orgene 유전자(roderived, ORF1b-Ns4, ORF1-0)에서 파생되었다. 모든 DNA올리고 오클레오타이드는 사용하기 전에 다시 무(無)의 물에 넣었다.

SAS-CoV-2에 설정된 ADT-PCR프라이머 프로브의 상대적 위치입니다. 세계 보건 기구(WHO)웹 사이트(http://www.who.int)에서 유전자와 프라이머 시퀀스를 검색했다. 아래의 엠플릭스 숫자는 SARS-CoV-2, GenBankMNP908947.3에 따른 게놈 위치입니다. The sets were published by China CDC (Orf1ab and N), Charité – universitätsmedizin berlin institute of virology in Germany (RdRp_SARSr and E), the University of Hong Kong (HKU-ORF1b_nsp14 and HKU-N), USA CDC (2019-nCoV_N1, N2, and N3), National Institute of Health in Thailand (WH-NIC N), and National Institute of Infectious Disease in Japan (NIID_ 2002-VGA_N). Orf1:openreadingframe1;RdA:RNA-dependentRNApolymerasegeneration;N/N64:비구 조적 단백질 14유전자;S:스파이크 단백질 유전자;E:포락선 단백질 유전자, N:nucleocapsid단백질 유전자 바이럴 RNA준비
감염 실험은 생물 무기 수준 3(BSL-3)실험실에서 수행되었다. 아프리카산 녹색 원숭이 신장 베로 세포(ATCCCCL-81)가 한국 질병 통제 예방 센터(CDC)에서 제공하는 임상 격리 SARS-CoV-2(BetaCoCorea/IgG/032020)에 감염됐다. 72시간 후, 성숙한 감염성 바이러스(바이러스 매체)를 포함하는 배양 배지를 수집하였고, 제조 업체의 지침에 따라 QImpmp바이러스 RNA추출 키트(독일, Qiagen, Hilden)를 사용하여 바이러스 RNA를 배양 배지에서 분리하였다.
시험관 내 transcriptRNA표준의 작성
The coding sequence of SARS-CoV-2 Envelope (E) protein, which cloned in pET21a plasmid was PCR amplified with T7 promoter primer (5’ – AATACGACTCACTATAG – 3’, Macrogen Inc., South Korea) and T7 terminator primer (5’ – GCTAGTTATTGCTCAGCGG – 3’, Macrogen) with AccuPower® PCR PreMix (-dye) kit (Bioneer Inc., South Korea). PCR제품은 MEGAscript™7TranscriptionKit(InvitrogenInc., CA, 미국)를 사용하여 체외 전사 템플릿으로 사용되었습니다. 시험관 내 transculatedRNA의 복사본 번호는 Quantus™Fluorometer(미국 WI, PromegaInc.)로 측정한 RNA농도에서 계산되었습니다. 표준화된 양의 체외 생성 RNA는 표준 곡선을 생성하기 위해 Eprimer와 LPT-PCR을 사용했습니다.
RoRd와 N의 적합한 BTT-PCR
추출된 핵산 샘플은 VTT-PCR에 의한 SARS-CoV-2의 비교 분석을 위해 테스트되었습니다. SA-COV-2의 Orf1및 N영역은 SA-COV-2특정 유전자의 대상 시퀀스로 사용되었습니다. 1X1단계 RT-PCR혼합물(WELLSBIOINC., SouthKorea)과 300nM의 프라이머와 탐침을 포함하는 20μL반응 용액에서 정제된 바이러스 RNA10μL가 증폭되었다. TDT-PCR은 CFX96터치 실시간 PCR검출 시스템(바이오-rad, 헤라클레스, CA, 미국)으로 수행되었습니다. 이 연구에 적용된 LPT-PCR조건은 다음과 같이 프로그래밍되었습니다. UNG배양, RT배양, 효소 활성화는 25°C에서 2분간, 55°C에서 10분간, 94°C에서 각각 3분간 연속으로 수행되었다. 그런 다음 열 순환은 94°C에서 15초(변성)동안, 60°C에서 45사이클 동안 30초 동안 수행되었습니다.
결과 및 토론.
PCR-PCR분석의 유효성 확인
Ct값은 음성 대조군에서 생성되지 않았으며 반응이 무균적으로 수행되었음을 나타냅니다. E유전자 프라이머-프로페어 세트의 표준 곡선은 또한 그에 따라 반응이 이루어졌음을 보여 주었다. 표준 곡선의 R2값은 0.999였으며, 계산된 증폭 효율은 101.6%였다. 이는 PCR-PCR반응이 최적의 상태로 수행되었음을 나타냅니다. 상등액과 세포 리사이트의 바이러스 농도는 Egene기반의 분석에 의해 결정되었다(표 2).

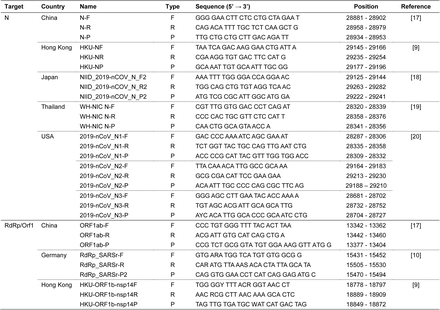
각 프라이머-프로브 세트를 사용하여 얻은 Ct값의 비교 분석 Rd/Or As As사이스
저 농도(15copies/38,38.97,36.85)의 Ctr값은 독일(Germany), HKU-orF-F10b-nsf(HongKong)및 ORF10/8탭(China)입니다(표 2). RdRp_SAR(독일)세트의 검사는 15카피/반응의 단일 반응에서 양성 신호를 3배로 나타냈다. HKU-orF17b-nsf(홍콩)및 ORF10/탭(중국)세트를 사용한 검사에서 1.5copies/reaction농도(데이터는 표시되지 않음)에서 양성 신호를 보여 주었습니다. Rd_SAR(독일), HKU-orF17b-nsf(홍콩)및 ORF10/8탭(중국)의 R2값은 각각 0.983,0.997 및 0.997이었습니다. 계산된 증폭 효율(독일)과 HKU-or17b-nsf(홍콩)및 ORF10/8탭(중국)은 각각 101.6%, 96.1%, 109.8%였습니다. 따라서, RoRd/Or/Orgene 유전자의 실험실 확인을 위해 ORASab(중국)세트가 권장될 수 있습니다.
엔 아사이
The Ct value of N (China), HKU-N (Hong Kong), NIID_2019-nCOV_N (Japan), WH-NIC N (Thailand), 2019-nCoV_N1, N2, and N3 (USA) from low concentration (15 copies/reaction) were 34.86, 35.43, 33.13, 38.13, 34.71, 33.14, and 33.09, respectively (Table 2). Ct값은 2019-LAN_N2, N3(미국)및 NIID_2019-ECOVE_N(일본)입니다. 이 값은 가장 민감한 세트로 간주될 수 있습니다. 다소 민감한 검사는 2002-VGA_N(미국)및 N(중국)을 기반으로 했습니다. 이러한 세트는 가장 민감한 세트보다 Ct값이 높지만, 저 농도(15copies/μl)의 Ct값은 여전히 컷오프 값(Ct<37)내에 있었습니다. WH-NICN(태국)세트는 다른 세트보다 덜 민감했습니다. 저 농도(15copies//μl)의 Ct값이 컷오프 값(Ct<38)에 근접했습니다. N(중국), HKU-N(홍콩), NIID_2019-ECF_N(일본), WH-NICN(태국), 2019-VGA_N1, N2및 N3(미국)의 R2값은 각각 0.989,0.980,0.987,0.986,0.951,0.991이었다. 계산된 증폭 효율은 각각 89.4,105.3,100.7,106.2,95.3,97.3,93.9,93.9, ID_2019-ECF_N(일본), WH-NICN(태국), WH-INC1, N2, N3(미국)입니다. 따라서, N유전자에 대한 TDT-PCR분석을 통해 SARS-COV-2를 실험실에서 확인하는 데는 2002-CCOP_N2, N3(미국)및 NIID_2002-ECOVE_N(일본)세트가 유리해야 한다.
결론들
이전에 다양한 프라이머-프로브 세트가 VTT-PCR분석을 통해 SARS-COV-2를 감지한 것으로 보고되었습니다. 검사의 민감도가 SARS-CoV-2감염의 초기 단계에서 의심스러운 환자를 확인하기에는 충분하지 않을 수 있다. 그럼에도 불구하고, SA-CoV-2의 분자 진단을 위한 현재의 PCR-PCR분석의 비교 결과는 없었습니다. 본 연구에서는 실험실 확인을 위해 LPT-PCR이 실험실 확인을 위해 다양한 프라이머-프로브 세트의 첫번째 비교 분석을 수행했다. Rd/OrOr 지역을 대상으로 하는 경우 ORF/ORF(중국)세트가 다른 세트보다 가장 민감할 수 있습니다. N부위의 민감한 VTT-PCR분석에는 2002-LAN_N2, N3(미국)및 NIID_2014-ECOVE_N(일본)세트를 권장할 수 있습니다. 따라서 SARS-CoV-2의 민감하고 신뢰할 수 있는 실험실 확인을 위해 ORF10탭(중국), 2002-VGA_N(미국)및 NIID_2019-ECOVN(일본)세트의 적절한 조합을 선택해야 한다.
감사의 말
우리는 한국 질병 통제 예방 센터의 병리학을 위한 국립 문화원에 임상적 SARS-CoV-2격리 장치를 제공해 준 것에 대해 감사합니다. 이 작업은 국가 과학 기술 연구회가 과학 기술부로부터 지원 받았다.
자료 출처 = https://www.krict.re.kr/news/bbs/view/tableid/news/id/92318
보도자료 | 한국화학연구원
□ 전 세계 연구진이 코로나19에 대항할 수 있는 연구를 진행하고 있는 가운데, 국내 연구진이 코로나19 치료용 항체 및 진단 개발에 활용할 수 있는 연구 결과를 발표했다. ◯ 한국화학연구원은 CEVI(신종 바이러스) 융합연구단은 기존에 알려진 사스 중화항체 2개와 메르스 중화항체 1개가 코로나19 스파이크 단백질에 결합할 수 있다는 결과를 예측했다. ◯ 스파이크 단백질은 코로나 바이러스가 세포 내로 침입할 때 활용되는 단백질이다. 연구진은 이 스파이크
www.krict.re.kr
자료출처 = https://www.biorxiv.org/content/10.1101/2020.02.25.964775v1
Comparative analysis of primer-probe sets for the laboratory confirmation of SARS-CoV-2
Coronavirus disease 2019 (COVID-19) is newly emerging human infectious diseases, which is caused by Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2, also previously known as 2019-nCoV). Within two months of the outbreak, more than 80,000 cases
www.biorxiv.org
코로나를 위해 힘써주시는 모든 분들에게 감사드립니다.
이번에도 대한민국의 힘을 보여줍시다~
코로나와 싸우고있는 모든분들 힘내세요
감사합니다.
'★사무업무★' 카테고리의 다른 글